基于生物信息学分析核仁纺锤体相关蛋白1在肝细胞癌中的表达及对临床预后的影响
DOI: 10.3969/j.issn.1001-5256.2021.07.024
Expression of nucleolar spindle-associated protein 1 in hepatocellular carcinoma and its effect on clinical prognosis: A bioinformatics analysis
-
摘要:
目的 探讨在肝癌中核仁纺锤体相关蛋白1(NUSAP1)的表达及其对临床预后的影响。 方法 从基因表达数据库(GEO)下载HCC芯片数据集GSE57957、GSE14520、GSE22058、GSE46444、GSE54236、GSE36376、GSE64041、GSE76297、GSE76427、GSE102079和癌症基因组图谱(TCGA)中的HCC RNA-seq数据,利用R 4.0软件分析NUSAP1在肝癌及癌旁组织的表达差异。应用t-分布邻域嵌入算法(tSNE)对单细胞测序数据的降维,分析NUSAP1在肝癌和癌旁组织中的差异表达。收集2018年1月—2019年11月在青岛大学附属医院进行同种异体肝移植术的42例乙型肝炎相关肝癌患者的癌组织及癌旁组织,收集其临床病理资料并分析与NUSAP1基因表达的相关性。计数资料2组间比较采用χ2检验。以Kaplan-Meier方法生存分析NUSAP1表达水平与总体生存率及无病间隔生存率之间的关系,运用log-rank进行检验,通过Graphpad prim 7使其结果可视化。 结果 NUSAP1在GEO数据库与TCGA数据库数据集中的肝癌组织中均高表达(P值均<0.001)。来源于肿瘤组织的细胞其NUSAP1高表达细胞明显高于癌旁组织细胞。NUSAP1在29例患者的肝癌组织中高表达,在13例患者的肝癌组织中低表达,NUSAP1在肝癌组织中高表达组及低表达组间,Child-Pugh分级、TNM分期、Okuda分期比较差异均有统计学意义(χ2值分别为5.469、6.836、4.617,P值均<0.05)。NUSAP1高表达的肝癌患者总体生存率与无病间隔生存率显著低于低表达的肝癌患者,NUSAP1低表达患者1、3、5生存率分别为92.96%、83.80%、76.76%,明显高于高表达的患者(76.76%、67.60%、64.78%),NUSAP1高表达的患者中位生存时间明显低于低表达的患者(61.7个月 vs 108.6个月)(P值均<0.05)。 结论 NUSAP1在肝癌细胞中高表达,可作为乙型肝炎肝癌患者不良预后的潜在标志物。 -
关键词:
- 癌,肝细胞 /
- 核仁纺锤体相关蛋白1 /
- 计算生物学
Abstract:Objective To investigate the expression of the nucleolar spindle-associated protein 1 (NUSAP1) gene in hepatocellular carcinoma (HCC) and its effect on clinical prognosis. Methods HCC microarray datasets GSE57957, GSE14520, GSE22058, GSE46444, GSE54236, GSE36376, GSE64041, GSE76297, GSE76427, and GSE102079 were downloaded from the Gene Expression Omnibus (GEO) database, and HCC RNA-Seq data were downloaded from The Cancer Genome Atlas (TCGA). R software 4.0 was used to investigate the difference in the expression of NUSAP1 between HCC tissue and adjacent tissue. The t-distributed stochastic neighbour embedding (t-SNE) method was used to perform dimensionality reduction of single-cell sequencing data and analyze the difference in the expression of NUSAP1 between HCC tissue and adjacent tissue. HCC tissue samples and adjacent tissue samples were collected from 42 patients with hepatitis B-related HCC who underwent liver transplantation in The Affiliated Hospital of Qingdao University from January 2018 to November 2019, and related clinical and pathological data were collected to analyze their correlation with the expression of the NUSAP1 gene. The chi-square test was used for comparison of paired data. The Kaplan-Meier method was used to analyze the correlation of the expression level of NUSAP1 with overall survival rate and disease-free survival, and the log-rank test was used for survival difference analysis; the results were visualized using Graphpad prim 7. Results NUSAP1 was highly expressed in HCC tissue in GEO and TCGA datasets (P < 0.001). The cells from HCC tissue had a significantly higher proportion of cells with high NUSAP1 expression than those from adjacent tissue. NUSAP1 showed high expression in HCC tissue of 29 patients and low expression in HCC tissue of 13 patients, and there were significant differences in Child-Pugh class, TNM stage, and Okuda stage between the high-expression group and the low-expression group (χ2=5.469, 6.836 and 4.617, all P < 0.05). The HCC patients with high expression of NUSAP1 had significantly lower overall survival rate and disease-free survival rate than those with low expression; compared with the patients with high expression, the patients with low expression had significantly higher 1-, 3-, and 5-year survival rates (92.96%/83.80%/76.76% vs 76.76%/67.60%/64.78%, P < 0.05), and the patients with high expression had a significantly shorter median survival time than those with low expression (61.7 months vs 108.6 months, P < 0.05). Conclusion NUSAP1 is highly expressed in cells from HCC tissue and may be used as a potential marker for poor prognosis of patients with hepatitis B-related HCC. -
肝癌作为消化系统最常见的恶性肿瘤之一,其发病率与致死率在各类肿瘤中一直位居前列[1]。我国每年肝癌新发病例数与死亡病例数约占全球总病例数的一半[2],其中肝细胞癌占绝大多数,因此本文主要讨论肝细胞癌。肝癌的发展,除因环境因素影响外,往往还与基因的突变或异常表达有关[3]。由于肝癌具有早期症状不明显、进展隐匿等特点,尽管在肝癌发现后可以进行手术为主的多种治疗方法,但总体预后不佳[4-6]。肿瘤发生与进展的关键在于正常的细胞周期被破坏,失去分化的能力,最终发生恶性增殖。因此,可以将抑制肝癌细胞的恶性增殖、恢复其正常的细胞进程作为治疗肝癌的潜在靶向。
核仁纺锤体相关蛋白1(nuclelar spindle-associated protein 1, NUSAP1)作为一种微管结合蛋白,在人体内高度保守,其作用在于调控纺锤体的正确装配以及染色体的形成,在有丝分裂过程中发挥不可或缺的作用[7-9]。在正常的组织中,NUSAP1的表达水平受到严格的调控,以防止细胞增殖异常。当NUSAP1表达异常增高时,会导致细胞有丝分裂进程无法终止,引起细胞的无限增殖以及遗传物质的不稳定。现有研究[10-16]已经证实,NUSAP1在多种肿瘤中表达异常增高,提示其密切参与肿瘤的发生与发展。
生物信息学分析在近几年越来越多的用于研究肿瘤进展过程中的基因表达变化[17]。通过对大量共享数据的挖掘、分析及解释,可以方便、快捷地发现具有表达差异的基因,找到疾病治疗的潜在靶标以及预后评估的可能标志物[18]。本研究通过对GEO及TCGA数据库公共数据的整理分析,对NUSAP1在肝癌组织及细胞亚群中的表达差异及其对预后的影响进行了初步的探索。
1. 资料与方法
1.1 数据收集与筛选
从TCGA数据库下载肝癌转录组信息及临床数据,选择临床信息完善的数据进行基因表达差异分析,以剔除肿瘤复发类型为非肝癌的R0切除术后患者的临床信息评估其预后。在GEO官网下载GSE57957、GSE14520、GSE22058、GSE46444、GSE54236、GSE36376、GSE64041、GSE76297、GSE76427、GSE102079共10个种属为Homo sapiens的肝癌数据集。在GEO官网下载单细胞测序数据集GSE149614。选取2018年1月—2019年11月在青岛大学附属医院进行同种异体肝移植术的随访信息完善、病理诊断明确、生物样本取材规范的乙型肝炎相关肝癌患者42例,统计其临床资料,包括性别、血型、年龄、BMI、血清AFP水平、MELD评分、Child-Pugh分级、肿瘤直径、肿瘤数目、TNM分期及Okuda分期等。
1.2 实时荧光定量PCR
对选取的42例临床样本利用Trizol法提取总RNA,并通过TaKaRa反转录试剂盒合成cDNA。利用TaKaRa PCR试剂盒进行实时荧光定量-PCR反应,以GAPDH为内参照,测定NUSAP1相对表达水平。GAPDH引物序列为:F:5′-CGGGCTCTCCAGAACATC-3′,R:5′-ATGACCTTGCCCACAGCCT-3′;NUSAP1引物序列为F:5′-ACCCTGACTCACAGCAGAATC-3′,R:5′-CAGCTTCTTGGTGCTCGTCT-3′。
1.3 伦理学审查
本研究方案经由青岛大学附属医院伦理委员会审批,批号:QYFYWZLL26046。
1.4 统计学方法
R 4.0软件用于所有数据集基因表达差异分析及单细胞测序数据的聚类分析。采用SPSS 21.0软件对数据进行统计分析。计数资料2组间比较采用χ2检验。以Kaplan-Meier方法生存分析NUSAP1表达水平与总体生存率及无病间隔生存率之间的关系,运用log-rank进行检验,通过Graphpad prim 7使其结果可视化。P<0.05为差异有统计学意义。
2. 结果
2.1 NUSAP1在肝癌组织及癌旁组织中的表达
为研究NUSAP1在肝癌组织中的表达,同时从GEO和TCGA中下载肝癌和癌旁组织基因表达数据。结果显示,NUSAP1在10个GEO肝细胞数据集中的肝癌组织中高表达,在TCGA数据库中也呈高表达(P值均<0.001)(附录1)。
2.2 NUSAP1在肝癌细胞中的表达
为避免肿瘤异质性对本研究的影响,从GEO中下载了肝细胞癌的单细胞测序数据集GSE149614并进行聚类分析。依据细胞标志物和组织来源的差异,将所有细胞分为来源于肿瘤组织的细胞与来源于癌旁组织的细胞。结果显示,来源于肿瘤组织的细胞其NUSAP1高表达细胞明显高于癌旁组织细胞(附录2)。
2.3 NUSAP1在临床肝癌样本中的表达
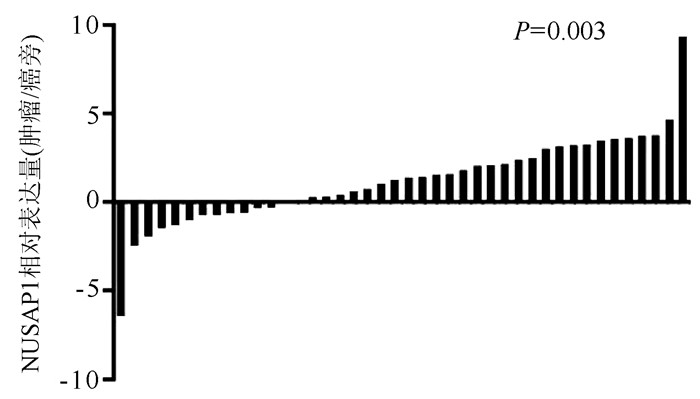
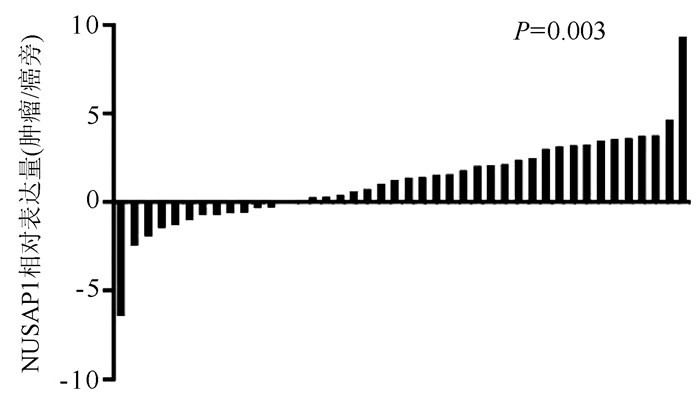
数据库的测序结果,通过实时荧光定量PCR检测42例患者肝癌和癌旁组织中NUSAP1的相对表达。与GEO和TCGA数据库分析结果一致,NUSAP1在29例患者的肝癌组织中高表达,在13例患者的肝癌组织中低表达(P=0.003)(图 1)。
2.4 NUSAP1表达水平与肝癌患者临床病理特征的关系
NUSAP1在肝细胞癌组织中高表达组及低表达组间,Child-Pugh分级、TNM分期、Okuda分期比较差异均有统计学意义(P值均<0.05)(表 1), 表明NUSAP1的表达水平与肝癌患者的病情严重程度有关。
表 1 NUSAP1表达水平与肝癌患者临床病理特征的关系项目 高表达(n=29) 低表达(n=13) χ2值 P值 性别(例) 0.022 0.88 男 24 11 女 5 2 血型(例) 0.628 0.89 O型 5 3 A型 11 5 B型 12 5 AB型 1 0 年龄 2.143 0.14 <50岁 16 4 ≥50岁 13 9 BMI 0.633 0.43 18.5~23.9 kg/m2 14 8 <18.5 kg/m2或≥24 kg/m2 15 5 AFP(例) 0.252 0.62 <20 ng/ml 11 6 ≥20 ng/ml 18 7 MELD评分(例) 0.032 0.86 <20分 17 8 ≥30分 12 5 Child-Pugh分级(例) 5.469 0.02 A级 7 8 B或C级 22 5 肿瘤数目(例) 0.001 0.97 单发 11 5 多发 18 8 TNM分期(例) 6.836 0.01 Ⅰ期 3 6 Ⅱ或Ⅲ期 26 7 Okuda分期(例) 4.617 0.03 Ⅰ 6 7 Ⅱ或Ⅲ 23 6 2.5 NUSAP1在肝细胞癌中高表达与预后的关系
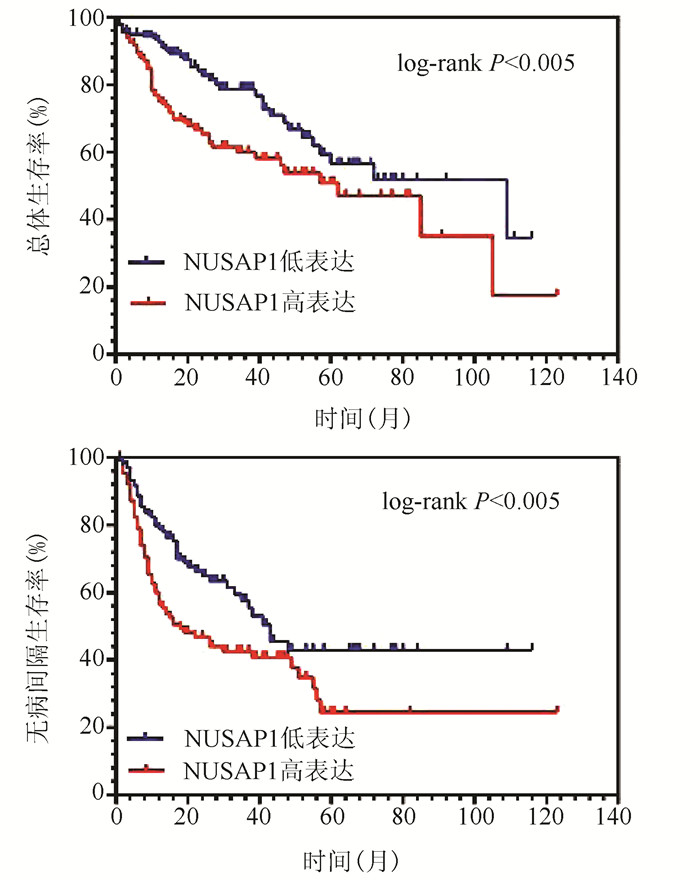
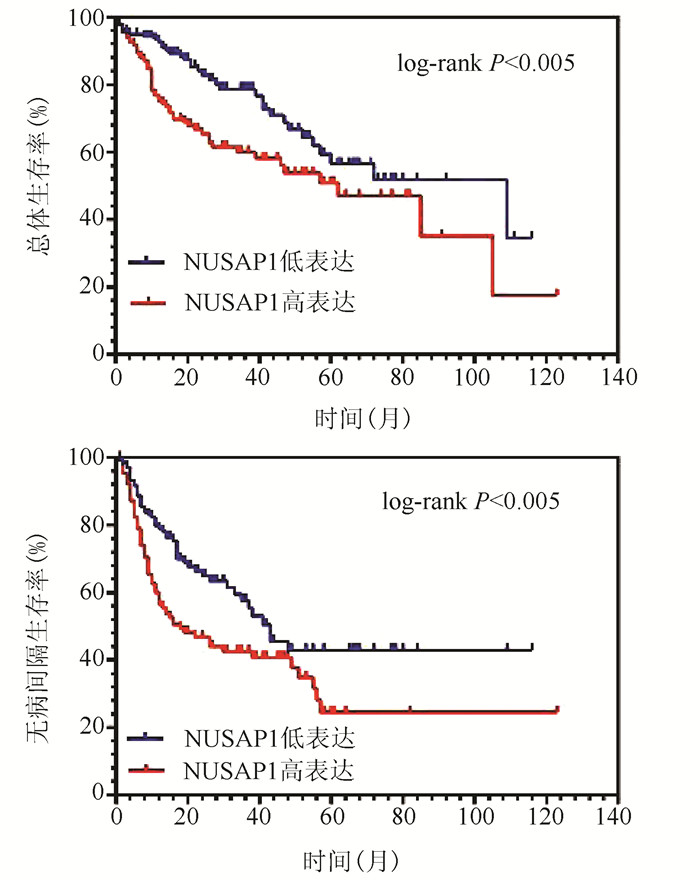
NUSAP1高表达的肝癌患者总体生存率与无病间隔生存率均显著低于低表达的肝癌患者(图 2),NUSAP1低表达患者1、3、5年生存率分别为92.96%、83.80%、76.76%,明显高于高表达的患者(76.76%、67.60%、64.78%)(χ2值分别为8.156、7.853、7.671, P值均<0.05),NUSAP1高表达的患者中位生存时间明显低于低表达的患者(61.7个月vs 108.6个月)(χ2=7.955, P<0.05)。
3. 讨论
肝癌的发生是一个复杂的过程,有多种细胞参与肝癌的发生与进展[19]。肝癌因其恶性程度高、易复发等特点,常见的治疗手段并不能显著改善肝癌患者的生存时间[20]。遗传因素作为限制肝癌患者预后的重要因素持续受到人们的关注。相比于传统的实验方法,随着生物信息学的发展以及肿瘤基因数据的共享,使得肿瘤相关基因的发现周期大为缩短,为研究肝癌发生发展机制,探寻潜在治疗靶点提供了重大便利。
NUSAP1作为有丝分裂中的一种关键蛋白,通过参与有丝分裂中纺锤体的形成得以调控细胞的增殖,其表达水平受到严格的调控,一旦NUSAP1表达异常升高,细胞将无限增殖,甚至影响遗传物质[9]。已有研究[14-15]证实,NUSAP1在多种肿瘤(如胶质母细胞癌、肾癌、乳腺癌等)中表达均显著提高,且均与预后有关,可以作为该型肿瘤的潜在标志物。本研究通过对大量GEO芯片数据及TCGA数据集的分析,证实NUSAP1在肝癌组织中的表达明显升高。通过对单细胞测序数据的分析,使NUSAP1高表达的肝癌细胞来源于肝癌组织中的推论更为明确,进一步证实肝癌细胞的无限增殖与NUSAP1表达升高的关系。由此推断,在肝脏因遗传因素或外界理化因素刺激后,NUSAP1表达出现异常增高,使肝细胞持续增殖,最终诱导癌变的产生。通过对TCGA数据库中患者预后信息的分析发现,NUSAP1的表达水平明显影响患者的预后,高表达组的患者总体生存率、无病间隔生存率均显著低于低表达组。进一步临床资料分析证实了上述结论,并发现NUSAP1在肝癌中的表达水平与Child-Pugh分级、TNM分期、Okuda分期显著相关,表明NUSAP1的表达升高使得肿瘤细胞增殖旺盛,使肝癌进展迅速,患者病情恶化加快,从而影响预后。综上所述,NUSAP1可以作为肝癌不良预后的新标志物并可以成为肝癌治疗的潜在新靶点。
综上所述,本研究将高通量基因芯片数据、单细胞测序数据的分析与现有临床样本PCR验证相结合,避免了单一研究方式的不足,结论更有说服力。研究验证了NUSAP1在肝癌细胞中的异常表达升高且与患者预后相关,为肝癌的靶向用药提供了新的潜在靶点。

-
表 1 NUSAP1表达水平与肝癌患者临床病理特征的关系
项目 高表达(n=29) 低表达(n=13) χ2值 P值 性别(例) 0.022 0.88 男 24 11 女 5 2 血型(例) 0.628 0.89 O型 5 3 A型 11 5 B型 12 5 AB型 1 0 年龄 2.143 0.14 <50岁 16 4 ≥50岁 13 9 BMI 0.633 0.43 18.5~23.9 kg/m2 14 8 <18.5 kg/m2或≥24 kg/m2 15 5 AFP(例) 0.252 0.62 <20 ng/ml 11 6 ≥20 ng/ml 18 7 MELD评分(例) 0.032 0.86 <20分 17 8 ≥30分 12 5 Child-Pugh分级(例) 5.469 0.02 A级 7 8 B或C级 22 5 肿瘤数目(例) 0.001 0.97 单发 11 5 多发 18 8 TNM分期(例) 6.836 0.01 Ⅰ期 3 6 Ⅱ或Ⅲ期 26 7 Okuda分期(例) 4.617 0.03 Ⅰ 6 7 Ⅱ或Ⅲ 23 6 -
[1] BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI: 10.3322/caac.21492. [2] KULIK L, EL-SERAG HB. Epidemiology and management of hepatocellular carcinoma[J]. Gastroenterology, 2019, 156(2): 477-491. e1. DOI: 10.1053/j.gastro.2018.08.065. [3] WANG SY, FENG LY, MENG ZQ. Bicluster and pathway enrichment analysis related to tumor progression of hepatocellular carcinoma[J]. Eur Rev Med Pharmacol Sci, 2015, 19(7): 1191-1197. http://europepmc.org/articles/PMC4237098 [4] BAATARKHUU O, GERELCHIMEG T, MUNKH-ORSHIKH D, et al. Epidemiology, genotype distribution, prognosis, control, and management of viral hepatitis B, C, D, and hepatocellular carcinoma in mongolia[J]. Euroasian J Hepatogastroenterol, 2018, 8(1): 57-62. DOI: 10.5005/jp-journals-10018-1260. [5] EL-SERAG HB. Epidemiology of viral hepatitis and hepatocellular carcinoma[J]. Gastroenterology, 2012, 142(6): 1264-1273. e1. DOI: 10.1053/j.gastro.2011.12.061. [6] FENG WG, WU W, GONG JP. Research progress on therapy of hepatocellular carcinoma[J]. Chin J Curr Adv Gen Surg, 2013, 16(7): 583-585. DOI: 10.3969/j.issn.1009-9905.2013.07.026.冯文贵, 吴伟, 龚建平. 肝细胞癌治疗的研究进展[J]. 中国现代普通外科进展, 2013, 16(7): 583-585. DOI: 10.3969/j.issn.1009-9905.2013.07.026. [7] RAEMAEKERS T, RIBBECK K, BEAUDOUIN J, et al. NuSAP, a novel microtubule-associated protein involved in mitotic spindle organization[J]. J Cell Biol, 2003, 162(6): 1017-1029. DOI: 10.1083/jcb.200302129. [8] PETRY S. Mechanisms of mitotic spindle assembly[J]. Annu Rev Biochem, 2016, 85: 659-683. DOI: 10.1146/annurev-biochem-060815-014528. [9] ZHOU Q, LEE KJ, KURASAWA Y, et al. Faithful chromosome segregation in Trypanosoma brucei requires a cohort of divergent spindle-associated proteins with distinct functions[J]. Nucleic Acids Res, 2018, 46(16): 8216-8231. DOI: 10.1093/nar/gky557. [10] LIU Z, GUAN C, LU C, et al. High NUSAP1 expression predicts poor prognosis in colon cancer[J]. Pathol Res Pract, 2018, 214(7): 968-973. DOI: 10.1016/j.prp.2018.05.017. [11] CHEN B, LI Y, WEI YH, et al. Expression and clinical significance of NUSAP1 in non-small cell lung cancer[J]. Int J Respir, 2017, 37(22): 1681-1684. DOI: 10.3760/cma.j.issn.1673-436X.2017.22.001.陈波, 李悦, 位云艳, 等. 非小细胞肺癌中NUSAP1的表达及临床意义[J]. 国际呼吸杂志, 2017, 37(22): 1681-1684. DOI: 10.3760/cma.j.issn.1673-436X.2017.22.001. [12] GAO S, YIN H, TONG H, et al. Nucleolar and spindle associated protein 1 (NUSAP1) promotes bladder cancer progression through the TGF-β signaling pathway[J]. Onco Targets Ther, 2020, 13: 813-825. DOI: 10.2147/OTT.S237127. [13] GE Y, LI Q, LIN L, et al. Downregulation of NUSAP1 suppresses cell proliferation, migration, and invasion via inhibiting mTORC1 signalling pathway in gastric cancer[J]. Cell Biochem Funct, 2020, 38(1): 28-37. DOI: 10.1002/cbf.3444. [14] GORDON CA, GULZAR ZG, BROOKS JD. NUSAP1 expression is upregulated by loss of RB1 in prostate cancer cells[J]. Prostate, 2015, 75(5): 517-526. DOI: 10.1002/pros.22938. [15] ZHANG X, PAN Y, FU H, et al. Nucleolar and spindle associated protein 1 (NUSAP1) inhibits cell proliferation and enhances susceptibility to epirubicin in invasive breast cancer cells by regulating cyclin D kinase (CDK1) and DLGAP5 expression[J]. Med Sci Monit, 2018, 24: 8553-8564. DOI: 10.12659/MSM.910364. [16] XIE Q, OU-YANG W, ZHANG M, et al. Decreased expression of NUSAP1 predicts poor overall survival in cervical cancer[J]. J Cancer, 2020, 11(10): 2852-2863. DOI: 10.7150/jca.34640. [17] LI Y, HUANG C, DING L, et al. Deep learning in bioinformatics: Introduction, application, and perspective in the big data era[J]. Methods, 2019, 166: 4-21. DOI: 10.1016/j.ymeth.2019.04.008. [18] SONG B, TANG JW, WANG B, et al. Identify lymphatic metastasis-associated genes in mouse hepatocarcinoma cell lines using gene chip[J]. World J Gastroenterol, 2005, 11(10): 1463-1472. DOI: 10.3748/wjg.v11.i10.1463. [19] ZHOU YL, LIU JY, YAO HW, et al. Increased expression of lncFOXC1 promotes liver cancer formation[J]. Chin J Clin Pharmacol Ther, 2019, 24(1): 27-31. DOI: 10.12092/j.issn.1009-2501.2019.01.005.周玉玲, 刘激扬, 姚红伟, 等. lncFOXC1表达增加促进肝癌发生[J]. 中国临床药理学与治疗学, 2019, 24(1): 27-31. DOI: 10.12092/j.issn.1009-2501.2019.01.005. [20] YUAN SX, ZHOU WP. Progress and hot spots of comprehensive treatment for primary liver cancer[J]. Chin J Dig Surg, 2021, 20(2): 163-170. DOI: 10.3760/cma.j.cn115610-20201211-00776.袁声贤, 周伟平. 原发性肝癌综合治疗的进展和热点[J]. 中华消化外科杂志, 2021, 20(2): 163-170. DOI: 10.3760/cma.j.cn115610-20201211-00776. 期刊类型引用(3)
1. 王锐,白皓天,李娅兰,杨婧. 基于生物信息学筛选并综合分析肝细胞癌关键基因. 海南医学院学报. 2022(21): 1634-1643 .  百度学术
百度学术2. 陈雪岩,乔建梁,李军,牛剑祥,赵建国,韩赛,孟兴凯. 小野寺预后营养指数对消化系统恶性肿瘤预后预测价值的研究进展. 中华消化外科杂志. 2022(10): 1390-1394 .  百度学术
百度学术3. 陈新宽,杨海燕,梁伟. NuSAP1、LDLR与局部晚期乳腺癌患者腋窝淋巴结转移的关系及其危险因素分析. 肿瘤代谢与营养电子杂志. 2022(06): 747-752 .  百度学术
百度学术其他类型引用(1)
-



 PDF下载 ( 2591 KB)
PDF下载 ( 2591 KB)


 下载:
下载:

 下载:
下载:
 百度学术
百度学术